IVF-PQ原理
IVF-PQ算法大致可以分为两部分:
- Product Quantizer
- Inverted File System
索引构建
数据分为训练集和数据集,有几个重要参数:
d: 向量的维度nb: 数据集向量个数nt: 参与训练的向量个数M: 指定将向量切分为几个子向量(注意d需要能被M整除)ncentroids: IVF划分多少个粗聚类簇nbits: PQ中子向量占据位数(bits),其决定了PQ子向量的聚类个数(1<<bits)
1. IVF聚类划分
将向量库中的向量做KMeans聚类,生成ncentroids个粗聚类簇。聚类中心向量存储于quantizer中。
2. PQ预训练
先将向量划分为M段,对每一段子向量做clustering。最终向量库的每段子向量都将被划分为1<<bits个(一般为256个)簇。为每个簇分配一个簇ID(以256为例,簇ID为0~255之间的数),这样一来向量库的向量大小就被压缩成M * nbits位。
搜索过程
搜索过程通过最大最小堆来维护搜索结果top N。
每当有查询向量进来,首先在IVF划分的ncentroids个粗聚类簇中选取top N个簇(这个N由参数nprobe参数配置),并计算查询向量与这些簇下面所有向量的聚类,计算距离的方法如下:
簇中的所有向量已经经过PQ的预训练,被压缩成N M个子向量,其中N为该簇中所包含的向量个数。拿之前的256举例,查询向量需要对M个子向量中的256个簇心向量分别计算距离并生成一张距离表(M 256)。如下图,可以看到表中保存了查询向量的M个子向量分别与256个簇心的距离。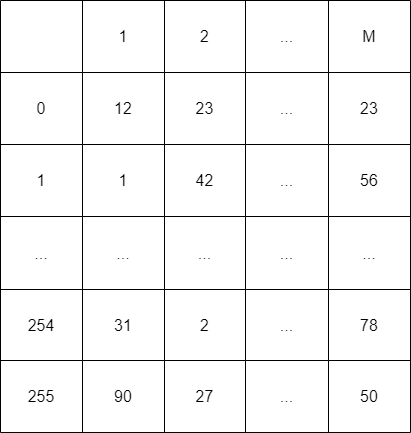
这样一来,查询向量与簇内向量中的距离就可以通过查询距离表来计算:例如簇内向量被量化为[cID1, cID2, ..., cIDm](cID表示簇ID),对于每段子向量的簇ID通过查询距离表就可以得到该子向量到达查询向量中对应子向量的距离,将每段子向量的距离相加即为查询向量到该簇内向量的距离。期间涉及了M 256次的向量计算以及N M次距离表查询。
IVF-PQ源码分析
源码地址:https://github.com/facebookresearch/faiss
train
数据训练
void IndexIVF::train(idx_t n, const float* x) {
// step1
train_q1(n, x, verbose, metric_type);
idx_t max_nt = train_encoder_num_vectors();
if (max_nt <= 0) {
max_nt = (size_t)1 << 35;
}
// 如果向量数太多(超过了max_nt),则对向量进行随机采样
TransformedVectors tv(
x, fvecs_maybe_subsample(d, (size_t*)&n, max_nt, x, verbose));
// step2 残差训练
if (by_residual) {
std::vector<idx_t> assign(n);
quantizer->assign(n, tv.x, assign.data());
std::vector<float> residuals(n * d);
quantizer->compute_residual_n(n, tv.x, residuals.data(), assign.data());
train_encoder(n, residuals.data(), assign.data());
} else {
train_encoder(n, tv.x, nullptr);
}
is_trained = true;
}1 train_q1
主要就是对训练向量进行粗聚类,其中最主要函数就是下面的train_encoded
1.1 train_encoded
对训练向量进行KMeans聚类,聚类结果保存于quantizer中。经过niter次迭代计算得到最终的ncentroids个聚类中心向量。
void Clustering::train_encoded(
idx_t nx,
const uint8_t* x_in, // 输入向量,原类型为float数组,这里转换成uint8_t数组
const Index* codec, // nullptr
Index& index, // quantizer
const float* weights) {
...
if (!codec) {
// Check for NaNs in input data. Normally it is the user's
// responsibility, but it may spare us some hard-to-debug
// reports.
const float* x = reinterpret_cast<const float*>(x_in);
for (size_t i = 0; i < nx * d; i++) {
FAISS_THROW_IF_NOT_MSG(
std::isfinite(x[i]), "input contains NaN's or Inf's");
}
}
...
// 存储每个向量属于哪个簇,以簇ID表示(簇中心向量 = centroids[d * 簇ID])
std::unique_ptr<idx_t[]> assign(new idx_t[nx]);
// 存储向量与选取聚类中心的距离
std::unique_ptr<float[]> dis(new float[nx]);
...
// 随机选取输入向量中的向量为centroids(d * nlist)赋值,然后计算距离...
centroids.resize(d * k);
std::vector<int> perm(nx);
rand_perm(perm.data(), nx, seed + 1 + redo * 15486557L);
for (int i = n_input_centroids; i < k; i++) {
memcpy(¢roids[i * d], x + perm[i] * line_size, line_size);
}
...
// 将centroids添加到quantizer中,这里涉及到sa_encode编码过程
index.add(k, centroids.data());
...
// KMeans聚类
float obj = 0;
for (int i = 0; i < niter; i++) {
// 计算聚类结果(dis, assign),这里会对输入向量和之前的
// centroids依次地进行KNNL2计算,计算时会将之前sa_encode后
// 数据再转成float数组。
index.search(
nx,
reinterpret_cast<const float*>(x),
1,
dis.get(),
assign.get());
...
// 用于存储每个簇中包含了多少的向量,维度为簇个数(nlist)
std::vector<float> hassign(k);
size_t k_frozen = frozen_centroids ? n_input_centroids : 0;
// 计算各个簇包含了多少个向量
compute_centroids(
d,
k,
nx,
k_frozen, // 不参与更新的centroids
x,
codec,
assign.get(),
weights,
hassign.data(),
centroids.data());
// 如果有的簇不包含向量,则随机选取一个包含较多向量的簇,从该簇中分一些向量。
// 做法就是将不包含向量的簇中心向量(centroids[d * ci])设置为被分裂的簇中心
// 向量(centroids[d * cj]), 然后再做一些微调整区分该两个簇的中心向量,再
// 将hassign[ci]设为hassign[cj]的一半,同时hassign[cj]减半完成分裂过程。
int nsplit = split_clusters(
d, k, nx, k_frozen, hassign.data(), centroids.data());
post_process_centroids();
// add centroids to index for the next iteration (or for output)
index.reset();
if (update_index) {
index.train(k, centroids.data());
}
index.add(k, centroids.data());
}
}关于距离的计算:IVF_PQ做KMeans粗聚类主要通过knnL2方法计算向量距离
2 train_encoder
训练乘积量化器
void IndexIVFPQ::train_encoder(idx_t n, const float* x, const idx_t* assign) {
pq.train(n, x);
if (do_polysemous_training) {
PolysemousTraining default_pt;
PolysemousTraining* pt =
polysemous_training ? polysemous_training : &default_pt;
pt->optimize_pq_for_hamming(pq, n, x);
}
if (by_residual) {
precompute_table();
}
}d = || x - y_C - y_R ||^2 (x为搜索向量,y_c为粗聚类簇心,y_r为PQ簇心) 上面个那个式子可以转化为一下形式:d = || x - y_C ||^2 + || y_R ||^2 + 2 * (y_C|y_R) - 2 * (x|y_R) ,其中|| y_R ||^2 + 2 * (y_C|y_R) 与x无关,这表示其可以提前在训练阶段计算出来,这样一来就可以大大提高搜索速度。然而存储|| y_R ||^2 + 2 * (y_C|y_R) 需要nlist * M * ksub (其中nlist为粗聚类数,M为子向量分段数,ksub为子向量聚类数)的空间,所以一般来说为了节省存储空间不会走precompute_table逻辑
2.1 ProductQuantizer::train
这里是在乘积量化器中对数据进行聚类,聚类方法依旧如上
void ProductQuantizer::train(size_t n, const float* x) {
if (train_type != Train_shared) {
// train_type默认为Train_default, 所以一般走这里的逻辑
train_type_t final_train_type;
final_train_type = train_type;
if (train_type == Train_hypercube ||
train_type == Train_hypercube_pca) {
if (dsub < nbits) {
final_train_type = Train_default;
printf("cannot train hypercube: nbits=%zd > log2(d=%zd)\n",
nbits,
dsub);
}
}
// 这里的dsub就是向量维度d除以M,表示每段子向量的维度。
// n为训练向量数
float* xslice = new float[n * dsub];
// 栈对象管理指针,离开作用域自动析构回收内存
ScopeDeleter<float> del(xslice);
for (int m = 0; m < M; m++) {
// 切分子向量拷贝到xslice
for (int j = 0; j < n; j++)
memcpy(xslice + j * dsub,
x + j * d + m * dsub,
dsub * sizeof(float));
// 构造聚类工具类对象,开始对子向量聚类
Clustering clus(dsub, ksub, cp);
// we have some initialization for the centroids
if (final_train_type != Train_default) {
clus.centroids.resize(dsub * ksub);
}
switch (final_train_type) {
case Train_hypercube:
init_hypercube(
dsub, nbits, n, xslice, clus.centroids.data());
break;
case Train_hypercube_pca:
init_hypercube_pca(
dsub, nbits, n, xslice, clus.centroids.data());
break;
case Train_hot_start:
memcpy(clus.centroids.data(),
get_centroids(m, 0),
dsub * ksub * sizeof(float));
break;
default:;
}
IndexFlatL2 index(dsub);
clus.train(n, xslice, assign_index ? *assign_index : index);
// 将聚类结果保存到ProductQuantizer::centroids(dsub * ksub * M)中
// 这里的ksub = 1<<nbits
set_params(clus.centroids.data(), m);
}
} else {
//
Clustering clus(dsub, ksub, cp);
if (verbose) {
clus.verbose = true;
printf("Training all PQ slices at once\n");
}
IndexFlatL2 index(dsub);
clus.train(n * M, x, assign_index ? *assign_index : index);
for (int m = 0; m < M; m++) {
set_params(clus.centroids.data(), m);
}
}
}Add
添加数据集
1 add_core_o
将数据集按照之前训练划分好的簇添加到倒排表中
void IndexIVFPQ::add_core_o(
idx_t n,
const float* x,
const idx_t* xids,
float* residuals_2,
const idx_t* precomputed_idx) {
idx_t bs = index_ivfpq_add_core_o_bs;
if (n > bs) {
for (idx_t i0 = 0; i0 < n; i0 += bs) {
idx_t i1 = std::min(i0 + bs, n);
if (verbose) {
printf("IndexIVFPQ::add_core_o: adding %" PRId64 ":%" PRId64
" / %" PRId64 "\n",
i0,
i1,
n);
}
add_core_o(
i1 - i0,
x + i0 * d,
xids ? xids + i0 : nullptr,
residuals_2 ? residuals_2 + i0 * d : nullptr,
precomputed_idx ? precomputed_idx + i0 : nullptr);
}
return;
}
InterruptCallback::check();
direct_map.check_can_add(xids);
FAISS_THROW_IF_NOT(is_trained);
double t0 = getmillisecs();
const idx_t* idx;
ScopeDeleter<idx_t> del_idx;
if (precomputed_idx) {
idx = precomputed_idx;
} else {
idx_t* idx0 = new idx_t[n];
del_idx.set(idx0);
quantizer->assign(n, x, idx0);
idx = idx0;
}
double t1 = getmillisecs();
uint8_t* xcodes = new uint8_t[n * code_size];
ScopeDeleter<uint8_t> del_xcodes(xcodes);
const float* to_encode = nullptr;
ScopeDeleter<float> del_to_encode;
if (by_residual) {
to_encode = compute_residuals(quantizer, n, x, idx);
del_to_encode.set(to_encode);
} else {
to_encode = x;
}
// 计算PQ距离表,这个过程将to_encode(原输入向量)转换成xcodes(量化后的向量)
pq.compute_codes(to_encode, xcodes, n);
//
size_t n_ignore = 0;
for (size_t i = 0; i < n; i++) {
idx_t key = idx[i];
idx_t id = xids ? xids[i] : ntotal + i;
if (key < 0) {
direct_map.add_single_id(id, -1, 0);
n_ignore++;
if (residuals_2)
memset(residuals_2, 0, sizeof(*residuals_2) * d);
continue;
}
uint8_t* code = xcodes + i * code_size;
size_t offset = invlists->add_entry(key, id, code);
if (residuals_2) {
float* res2 = residuals_2 + i * d;
const float* xi = to_encode + i * d;
pq.decode(code, res2);
for (int j = 0; j < d; j++)
res2[j] = xi[j] - res2[j];
}
direct_map.add_single_id(id, key, offset);
}
ntotal += n;
}1.1 compute_code
按照之前设定的nbit(推荐为8)进行量化编码,编码过程可以概括为将输入向量的每段分别与之前训练好各段的256(nbits=8)个聚类中心计算距离,在找出距离最近的
void ProductQuantizer::compute_code(const float* x, uint8_t* code) const {
switch (nbits) {
case 8:
faiss::compute_code<PQEncoder8>(*this, x, code);
break;
case 16:
faiss::compute_code<PQEncoder16>(*this, x, code);
break;
default:
faiss::compute_code<PQEncoderGeneric>(*this, x, code);
break;
}
}
// 计算单个向量的pq距离
void compute_code(const ProductQuantizer& pq, const float* x, uint8_t* code) {
std::vector<float> distances(pq.ksub);
// 构造一个encode(8bit)
PQEncoder encoder(code, pq.nbits);
// 分M段计算
for (size_t m = 0; m < pq.M; m++) {
const float* xsub = x + m * pq.dsub;
uint64_t idxm = 0;
//离找到该段最近的簇ID(idxm),则该段子向量可以用该簇ID(nbit)来表示
idxm = fvec_L2sqr_ny_nearest(
distances.data(),
xsub,
pq.get_centroids(m, 0),
pq.dsub,
pq.ksub);
encoder.encode(idxm);
}
}